| О журнале | Редколлегия | Редсовет | Архив номеров | Поиск | Авторам | Рецензентам | English |
Использование молекулярного докинга по ферменту гидролаза E.Coli (S.Aureus) в исследовании связи «структура-активность» в ряду замещенных амидов и гидразидов N-ароилантраниловых кислот
Автор для корреспонденции: Андрюков Константин Вячеславович; e-mail: k_andrukov@mail.ru. Финансирование. Исследование не имело спонсорской поддержки. Конфликт интересов. Авторы заявляют об отсутствии конфликта интересов. Аннотация Данная статья посвящена использованию молекулярного докинга по ферменту гидролаза E.Coli (S.Aureus) в исследовании связи "структура-активность" в ряду замещенных амидов и гидразидов N-ароилантраниловых кислот. Для построения модели "структура-активность" использовали результаты молекулярного докинга по ферменту пептид деформилаза (гидролаза) E.Coli и S.Aureus производных амидов и гидразидов N-ароил антраниловых кислот. При проведении докинга использовали ферменты гидролаза E.Coli (PDB ID code: 1LRU) и S.Aureus (PDB ID code: 1Q1Y), структуры которых были получены из базы данных RCSB Protein Data Bank. Перед проведением молекулярного докинга выполнена оптимизация и квантово-химический расчёт структур исследуемых соединений полуэмпирическим методом РМ3 с использованием программы Gaussian 03. Определены значимые параметры: суммарные значения напряженности электрического поля Σ(Е), потенциал Σ(φ) и абсолютная величина заряда Σ(|q|) на атомах кислорода, азота, углерода и водорода. Провели теоретический расчёт физико-химических дескрипторов исследуемых соединений: констант липофильности (logPрассч), констант кислотности (pKaрассч) и основности (pKврассч). Моделирование лиганд-рецепторных взаимодействий осуществляли программой AutoDock 4.0 в составе программного комплекса MGL Tools 1.5.6, с использованием Ламарковского генетического алгоритма. В результате проведенного докинга по ферментам гидролаза E.Coli и S.Aureus, получены скоринговые функции: энергия связывания (Binding energy (BeE.Coli и BeS.Aureus)), межмолекулярная энергия (Intermolecular energy (ImeE.Coli и ImeS.Aureus)) и константа ингибирования (KiE.Coli и KiS.Aureus), характеризующие взаимодействие лиганда с рецептором. Выполнены исследования зависимости противомикробной активности (ПМА) от скоринговых функций и физико-химических дескрипторов. Составлены модели "структура-активность" проведением множественного линейного регрессионного анализа с использованием программы Statistica 6. Получено четыре корреляционных уравнения связывающих скоринговые функции и физико-химические дескрипторы с противомикробной активностью с использованием 20 соединений. Выполнена проверка составленных моделей на примере 4 соединений. Линейные зависимости ПМАрассч. от экспериментальных значений ПМАэксп. (МПКэксп. (E.Coli) и МПКэксп.(S.Aureus)) для уравнений 2 и 4, из четырех составленных, с коэффициентами корреляции (Rпрогн. (2)= 0,961 и Rпрогн. (4)= 0,911), показывают высокую степень связи ПМАрассч. c ПМАэксп.. Ключевые слова амид, гидразид, антраниловая кислота, молекулярный докинг, противомикробная активность, квантово-химические параметры, структура-активность doi: 10.29234/2308-9113-2019-7-4-94-109 Для цитирования: Андрюков К. В., Коркодинова Л. М. Использование молекулярного докинга по ферменту гидролаза E.Coli (S.Aureus) в исследовании связи «структура-активность» в ряду замещенных амидов и гидразидов N-ароилантраниловых кислот. Медицина 2019; 7(4): 94-109 Введение Моделирование взаимодействия с организмом с целью проведения скрининга с использованием молекулярного докинга и полученных на его основе соотношений "структура-активность", позволяет проводить целенаправленный поиск соединений с выбранным видом фармакологической активности. Использование молекулярного докинга возможно в том случае если известна мишень, с которой взаимодействует исследуемый лиганд. Перед проведением скрининга методом молекулярного докинга важно узнать фермент, воздействие на который, приводит к появлению у исследуемого лиганда анализируемой биологической активности. Антраниловая кислота принимает участие в широком спектре биохимических реакций, используется в качестве одного из ключевых реагентов в полусинтетическом методе получения ряда антибиотиков, как например, кальцимицина, проявляющего противомикробную активность [16]. Производные антраниловой кислоты, а именно амиды и гидразиды, относятся к классу биологически активных соединений, обладающих широким спектром фармакологической активности: противомикробной [10,13,17], противовоспалительной [8,15] и анальгетической [11]. Проведение моделирования по противомикробной активности является актуальным при проведении целенаправленного поиска веществ с выраженной противомикробной активностью [12,18]. Сочетание молекулярного докинга с физико-химическими дескрипторами, позволяет учитывать в проводимых исследованиях физико-химические свойства веществ, увеличивая качество модели, как например, модель "структура-противовоспалительная активность" [2]. Цель работы заключается в исследовании связи "структура-активность" с использованием молекулярного докинга по ферменту гидролаза E.Coli (S.Aureus) в ряду замещенных амидов и гидразидов N-ароилантраниловых кислот (20 соединений). 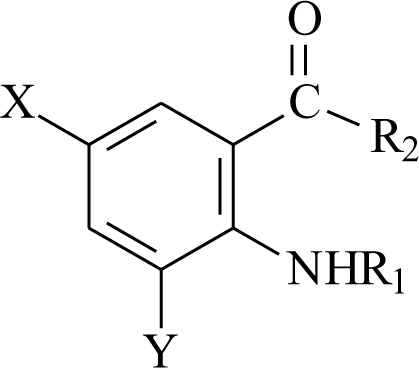 X=Br, Y=H, R1 = H, R2 = NH2 (1); X=Br, Y=H, R1 = CO 2–COOHC6Cl4, R2 = NH2 (2); X=Br, Y=H, R1 = CO 3–NO2 C6H4, R2 = NH2 (3); X=Br, Y=H, R1 = CO 4–CH3 C6H4, R2 = NH2 (4); X=Cl, Y=H, R1 = CO 4–Cl C6H4, R2 = NH CH2C6H5 (5); X=Br, Y=H, R1 = CO C6H5, R2 = NH C6H5 (6); X=Br, Y=H, R1 = CO 2-фурил, R2 = NH NHCO(2-фурил) (7); X=Br, Y=H, R1 = CO C6H5, R2 = NH CH2CH2OH (8); X=Br, Y=H, R1 = CO 4–Cl C6H4, R2 = NH2 (9); X=Cl, Y=H, R1 = CO 4–Cl C6H4, R2 = NH 2–CH3 C6H4 (10); X=Br, Y=H, R1 = CO 4–NO2 C6H4, R2 = NH C6H5 (11); X=Br, Y=H, R1 = CO 2-фурил, R2 = NH NHCOCH2Cl (12); X=Br, Y=H, R1 = CO C6H5, R2 = NH2 (13); X=Br, Y=H, R1 = CO 2–OCH3 C6H4, R2 = NH2 (14); X=Br, Y=H, R1 = CO 4–NO2 C6H4, R2 = NH CH3 (15); X=Br, Y=H, R1 = CO 4–NO2 C6H4, R2 = морфолил (16); X=I, Y=H, R1 = CO 4–CH3 C6H4, R2 = морфолил (17); X=I, Y=H, R1 = CO C6H5, R2 = NH NHCO-2-фурил (18); X=I, Y=H, R1 = CO C6H5, R2 = NH NH COCH2CH(CH3)2 (19); X=Br, Y=Br, R1 = CO C6H5, R2 = NH С6H11 (циклогексил) (20). Материал и методы исследования Оптимизацию структуры и расчёт квантово-химических дескрипторов исследуемых замещенных амидов и гидразидов N-ароилантраниловых кислот проводили с использованием программы Gaussian 03 полуэмпирическим методом РМ3, с полной оптимизации геометрии молекул. Бактериостатическую активность по отношению к штаммам S.Aureus ATCC 6538-P и E.Coli ATCC 25922 определяли методом двухкратных серийных разведений в жидкой питательной среде [7]. В качестве растворителя соединений использовали диметилсульфоксид. Посевы производили в мясопептонный бульон (pH 7,0) с различной концентрацией испытуемых соединений. Микробная нагрузка составила 250 000 микробных тел на 1 мл. Моделирование лиганд-рецепторных взаимодействий осуществляли программой AutoDock 4.0 в составе программного комплекса MGL Tools 1.5.6, с использованием Ламарковского генетического алгоритма, который позволяет воспроизводить строение комплексов более точно [14]. При проведении молекулярного докинга использовали трёхмерные модели молекул фермента пептид деформилаза (гидролаза) E.Coli и S.Aureus, информация о которых получена из базы данных RCSB Protein Data Bank: гидролаза E.Coli (PDB ID code: 1LRU [9]) и гидролаза S.Aureus (PDB ID code: 1Q1Y [20]). Изначально, все молекулы воды были удалены из структуры белка. Файлы рецептора и лигандов были конвертированы в формат PDBQT-файла, с добавлением недостающих атомов водорода и частичных атомных зарядов по методу Гастейгера. Результаты Структуры всех лигандов были построены и оптимизированы с использованием программы Gaussian 03 полуэмпирическим методом РМ3 с полной оптимизацией геометрии молекул, и затем были конвертированы в 3D-формат (.pdb) с помощью программы ChemBio3D Ultra 12,0. В результате проведенных квантово-химических расчётов программой Gaussian 03, получены квантово-химические дескрипторы: суммарные значения напряженности электрического поля Σ(Е), потенциала Σ(φ) и заряда в модуле Σ (|q|) на атомах углерода, кислорода, азота и водорода для всей молекулы. С использованием полученных квантово-химических дескрипторов: суммарная напряженность на атомах углерода (C(E)) и азота (N(E)), суммарный потенциал по атомам углерода (C(φ)), кислорода (O(φ)) и азота (N(φ)), и суммарный заряд в модуле по атому кислорода (O(|q|)), провели теоретический расчёт физико-химических дескрипторов исследуемых соединений: констант липофильности (log Pрассч) [3], констант кислотности (pKaрассч) [1] и основности (pKврассч) [1] (таблица 1). Значения констант липофильности лежат в интервале от 2,12 у соединения 1 до 3,90 (соединение 14). Константы кислотности, характеризующие ионизацию, рассчитанные значения которых находятся в пределах от 6,49 (соединение 2) до 9,58, для соединения 16. Константы основности, находятся в пределах от 11,82 у соединения 16 до 13,16, для соединения 5. Таблица 1. Квантово-химические дескрипторы, log Pрассч, pKaрассч и pKврассч замещенных амидов и гидразидов N-ароилантраниловых кислот 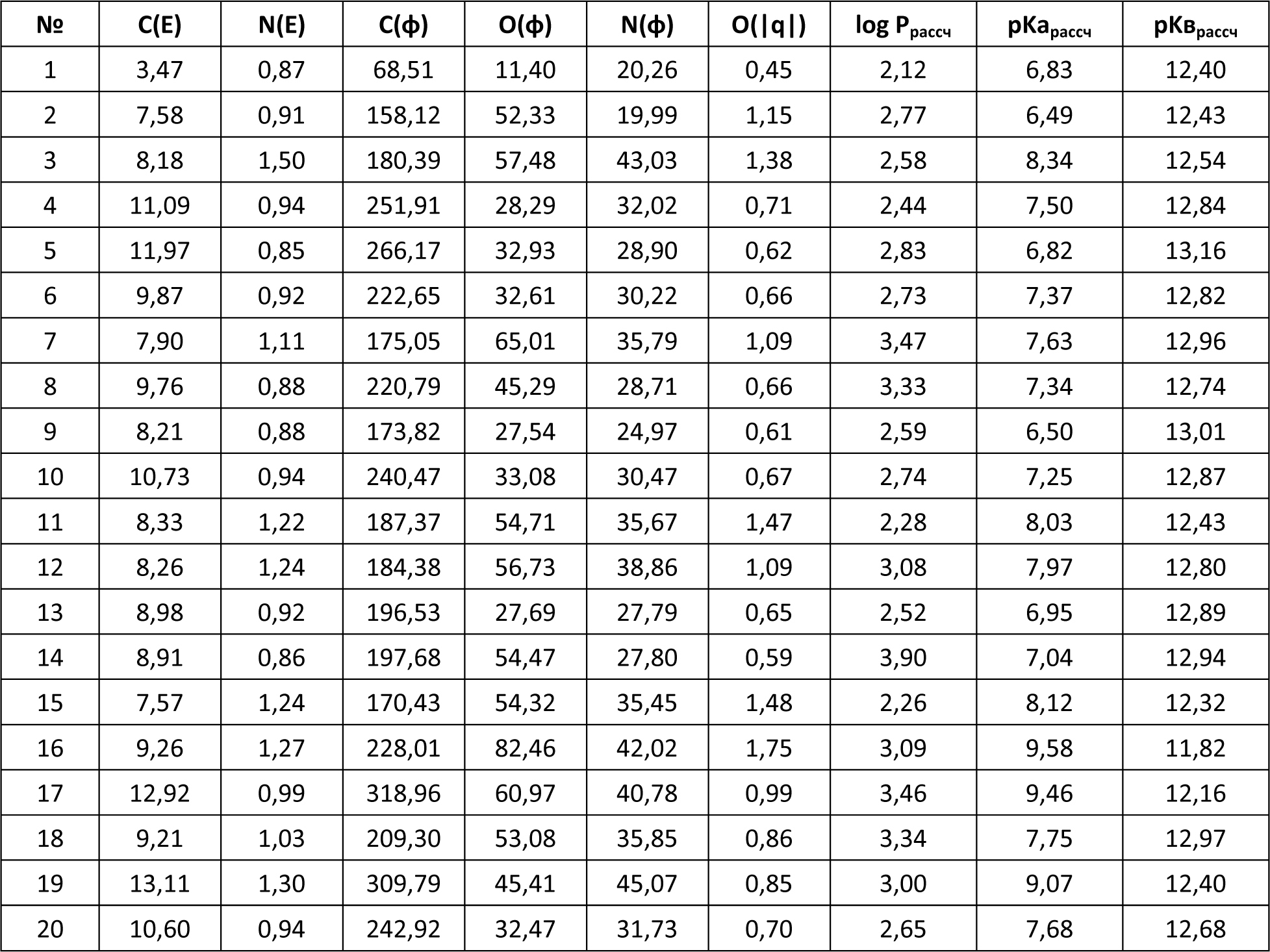 Для 20 соединений из ряда замещенных амидов и гидразидов N-ароилантраниловых кислот, была экспериментально определена противомикробная активность (ПМАэксп.) в отношении E.Coli и S.Aureus (таблицы 2 и 3). Бактериостатическую активность по отношению к штаммам S.Aureus ATCC 6538-P и E.Coli ATCC 25922 определяли методом двухкратных серийных разведений в жидкой питательной среде [7]. В качестве растворителя соединений использовали диметилсульфоксид. Посевы производили в мясопептонный бульон (pH 7,0) с различной концентрацией испытуемых соединений. Микробная нагрузка составила 250 000 микробных тел на 1 мл. Через 18-20 ч выдержки контрольных пробирок в термостате при температуре 370 С фиксировали результат. Для всех исследуемых 20 соединений определяли минимальные подавляющие концентрации (МПК) (таблицы 2 и 3) [4,5,6]. Установлено, что большинство изучаемых амидов обладает слабой бактериостатической активностью (таблицы 2 и 3), только соединение 2, по структуре амид тетрахлорфталил 5-бромантраниловой кислоты, проявило бактериостатическую активность равную 15,6 мкг/мл, в отношении обоих штаммов. Перед проведением множественного линейного регрессионного анализа, с целью получения равномерной выборки наблюдаемых значений, результат по противомикробной активности МПК (мкг/мл) переводили в логарифм: log (1/МПК), который затем использовали при составлении уравнений и дальнейшей валидации полученных моделей. Моделирование лиганд-рецепторных взаимодействий осуществляли программой AutoDock 4.0 в составе программного комплекса MGL Tools 1.5.6, с использованием Ламарковского генетического алгоритма, который позволяет воспроизводить строение комплексов более точно [14]. При проведении молекулярного докинга мы использовали трёхмерные модели молекул фермента пептид деформилаза (гидролаза) E.Coli и S.Aureus, информация о которых получена из базы данных RCSB Protein Data Bank: гидролаза E.Coli (PDB ID code: 1LRU [9]) и гидролаза S.Aureus (PDB ID code: 1Q1Y [20]). Изначально, все молекулы воды были удалены из структуры белка. Файлы рецептора и лигандов были конвертированы в формат PDBQT-файла, с добавлением недостающих атомов водорода и частичных атомных зарядов по методу Гастейгера. В процедуре докинга с координатами точек (60×60×60) вокруг каждого моделируемого участка, по ферменту гидролаза E.Coli при построении Grid-карт за центр были взяты координаты лиганда (x = –35,18, y = 28,54, z = 64,81), а для фермента гидролаза S.Aureus координаты лиганда (x = –18,26, y = 141,93, z = 49,21). В результате получено 20 Grid-карт по параметрам для исследуемых соединений. Оценку качества позиционирования характеризовали величиной RMSD, представляющей собой среднеквадратичное отклонение положения лиганда после докинга от его начального положения. В качестве активного сайта фермента гидролаза E.Coli был принят участок макромолекулы, содержащий аминокислоты (GLU133, LEU91, LEU125, GLY89, GLU87, ILE44) [9] и для фермента гидролаза S.Aureus, содержащий аминокислоты (GLU155, VAL151, VAL59, GLN65) [20]. Отбор значимых конформаций осуществляли по минимальному значению среднеквадратичного отклонения ∆RMSD от молекулы антибиотика актинонина, использованного в качестве исходной молекулы при построении структур. Результат проведенного молекулярного докинга по ферментам гидролаза E.Coli и S.Aureus представлен в таблицах 2 и 3 в виде скоринговых функций: энергия связывания (Binding energy (BeE.Coli и BeS.Aureus)), межмолекулярная энергия (Intermolecular energy (ImeE.Coli и ImeS.Aureus)) и константа ингибирования (KiE.Coli и KiS.Aureus), характеризующие взаимодействие лиганда с рецептором (гидролаза E.Coli и S.Aureus). Для соединения 2 с ПМА равной 15,6 мкг/мл приведены диаграммы взаимодействия с гидролазой E.Coli (рисунок 1) и S.Aureus (рисунок 2), включающие водородные связи и гидрофобные взаимодействия по аминокислотам. Таблица 2. Скоринговые функции и МПК(E.Coli)эксп., log(1/МПК)(E.Coli)эксп. замещенных амидов и гидразидов N-ароилантраниловых кислот (20 соединений) 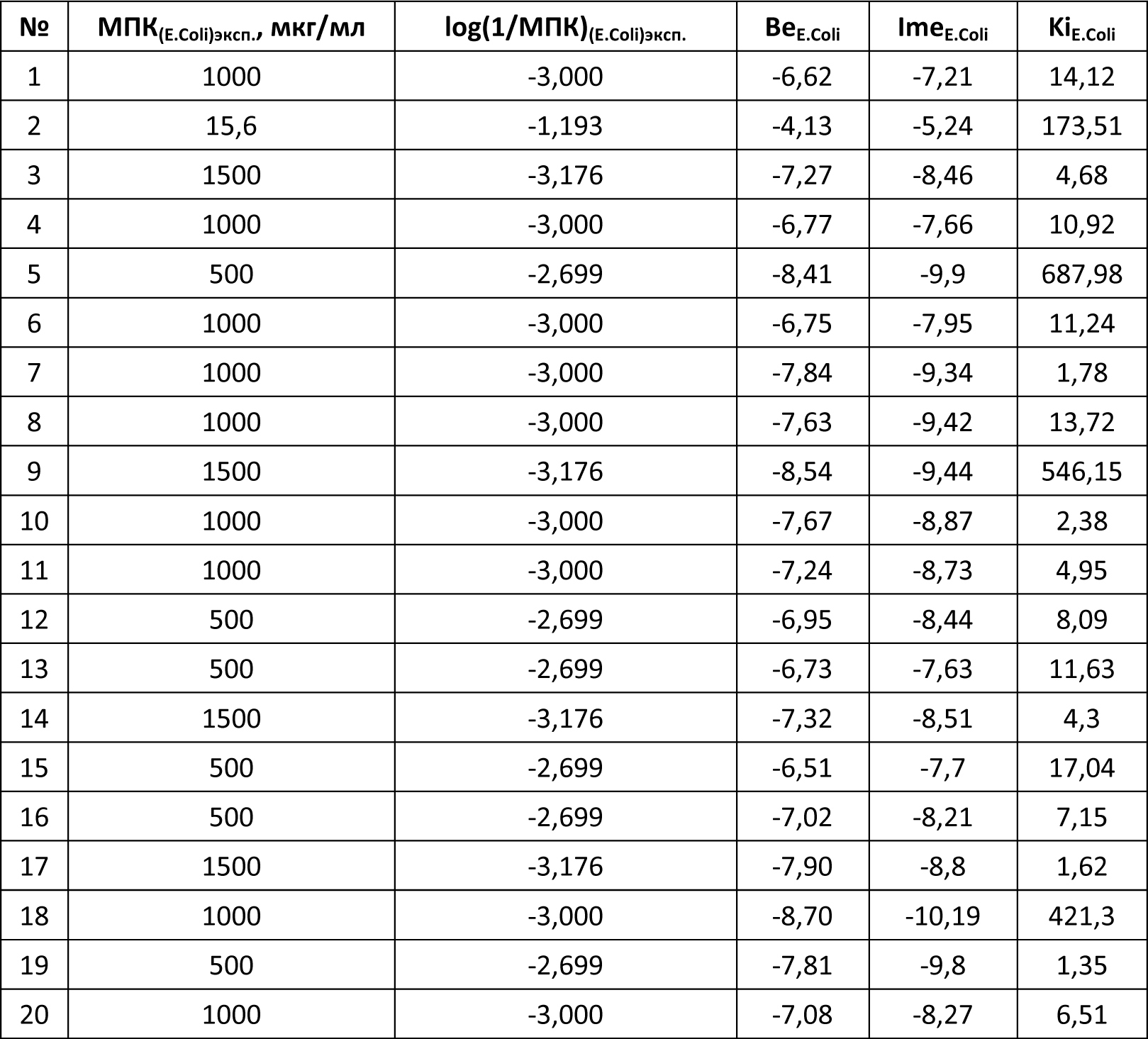 Таблица 3. Скоринговые функции и МПК(S.Aureus)эксп., log(1/МПК)(S.Aureus)эксп. замещенных амидов и гидразидов N-ароилантраниловых кислот (20 соединений) 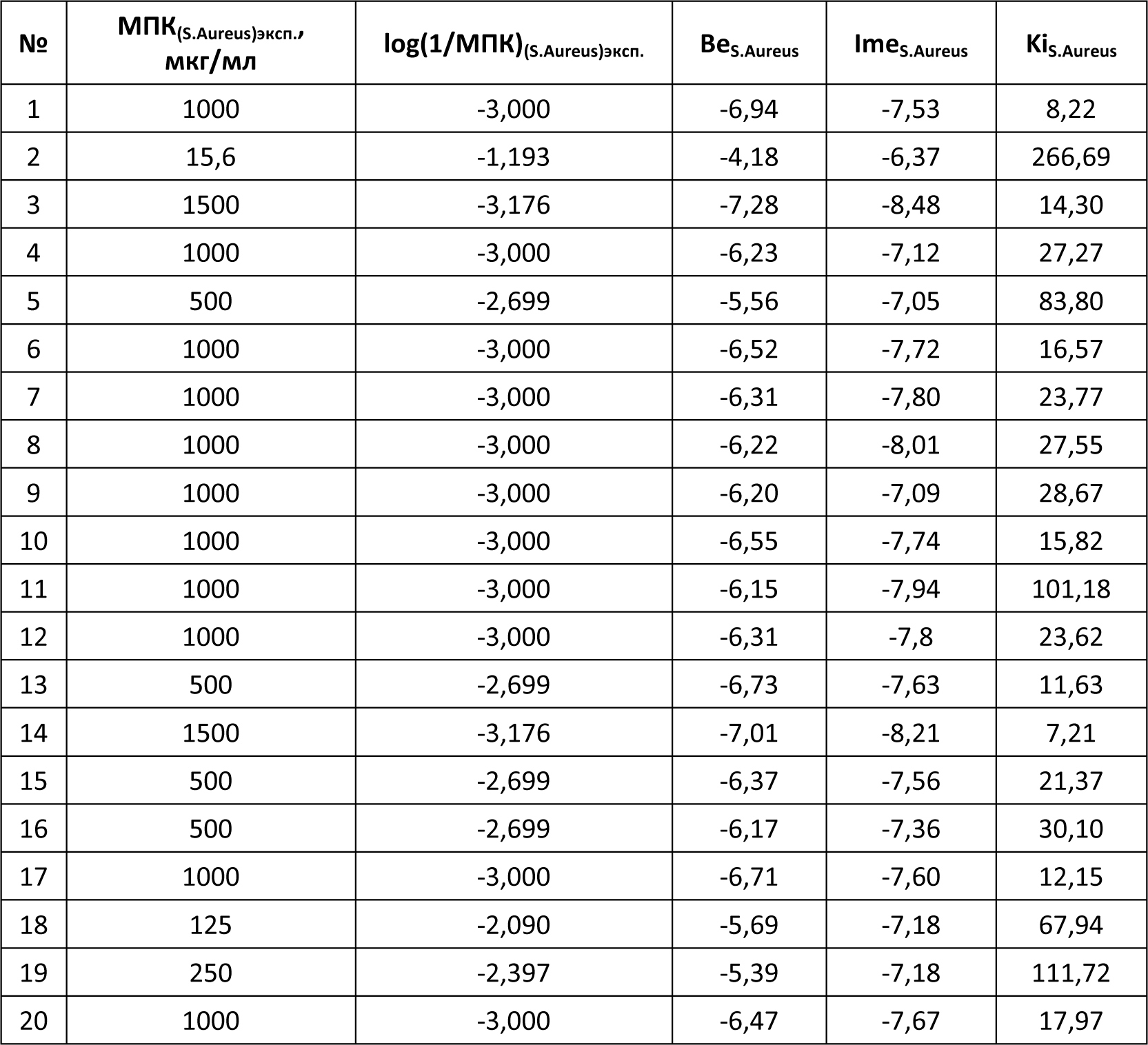 По атому брома и бензольному кольцу антраниловой кислоты происходит гидрофобное взаимодействие соединения 2 с активным участком фермента – гидролазы E.Coli (рисунок 1) содержащим аминокислоты изолейцин (ILE44) и лейцин (LEU125). Также образуется межмолекулярная водородная связь с аминокислотой аргинин (ARG97) по карбоксилат иону карбоксильной группы заместителя в NH-ацильном фрагменте. Соединение 2 взаимодействует с гидролазой S.Aureus гидрофобными фрагментами: ароматическим кольцом ядра антраниловой кислоты, атомом брома и атомами хлора в NH-ацильном фрагменте, водородные связи при этом не образуются. Заместитель при NH-ацильной группе, содержащий атомы хлора, вступает в гидрофобное взаимодействие с аминокислотами активного участка гидролазы S.Aureus – валин (VAL59 и VAL151). Рис. 1. Комплекс соединения 2 с активным участком гидролазы E.Coli. Изображение получено с использованием программы LigandScout. 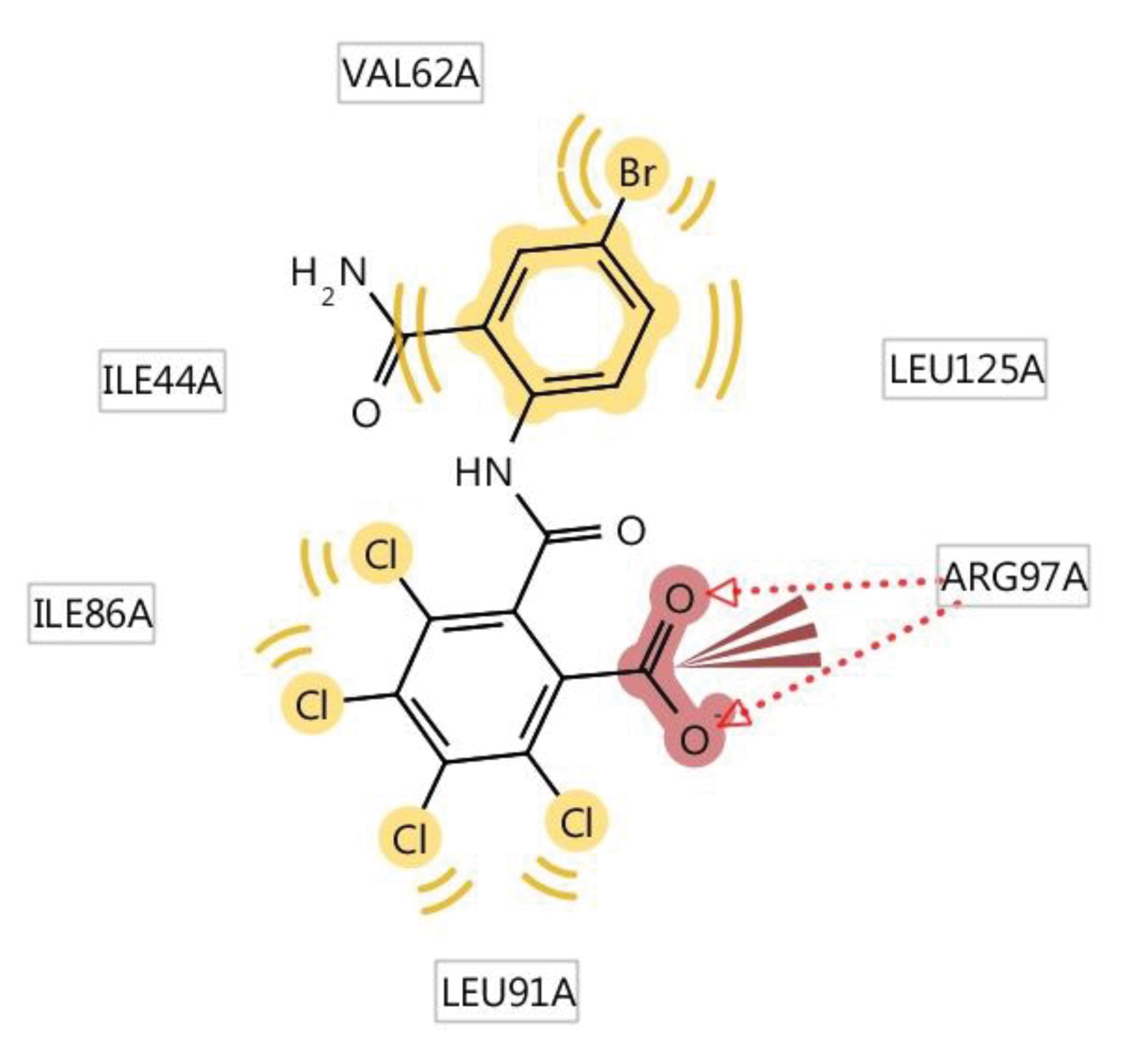 Рис. 2. Комплекс соединения 2 с активным участком гидролазы S.Aureus. Изображение получено с использованием программы LigandScout. 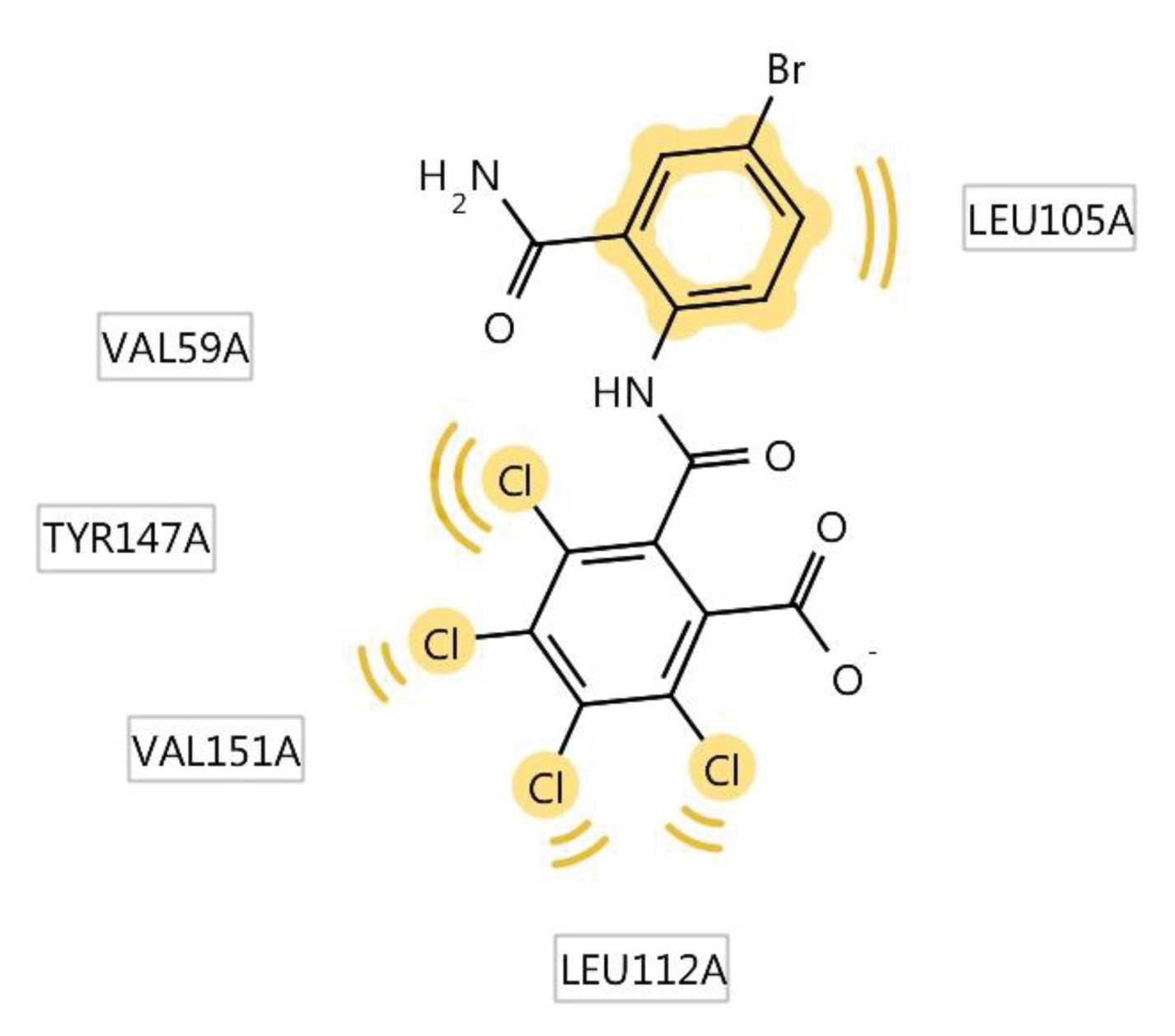 Обсуждение результатов С целью составления модели "структура-активность", проведен множественный линейный регрессионный анализ с использованием программы Statistica 6, зависимости log(1/МПК)E.Coli и log(1/МПК)S.Aureus от констант липофильности (logPрассч), ионизации (pKaрассч и pKврассч) и скоринговых функций по ферментам гидролаза E.Coli (BeE.Coli, ImeE.Coli, KiE.Coli) и S.Aureus (BeS.Aureus, ImeS.Aureus, KiS.Aureus). Всего было сгенерировано свыше 43 уравнений регрессии, из которых методом пошагового включения параметров с использованием программы Statistica 6, были отобраны 4 уравнения (таблица 4) зависимости экспериментально определенной ПМА (log(1/МПК)E.Coli и log(1/МПК)S.Aureus) от logPрассч, pKaрассч, BeE.Coli, ImeE.Coli, KiE.Coli, BeS.Aureus, ImeS.Aureus, KiS.Aureus. Установлено, что полученные уравнения имеют наибольшие значения коэффициента корреляции (R), критерия Фишера (F) и критерия оценки методом перекрестного контроля исключением по одному (Q2LOO), и минимальное значение среднеквадратичной ошибки (S) (таблица 4). Таблица 4. Уравнения множественной регрессии зависимости log(1/МПК)E.Coli и log(1/МПК)S.Aureus от logPрассч, pKaрассч, BeE.Coli, ImeE.Coli, KiE.Coli, BeS.Aureus, ImeS.Aureus, KiS.Aureus. 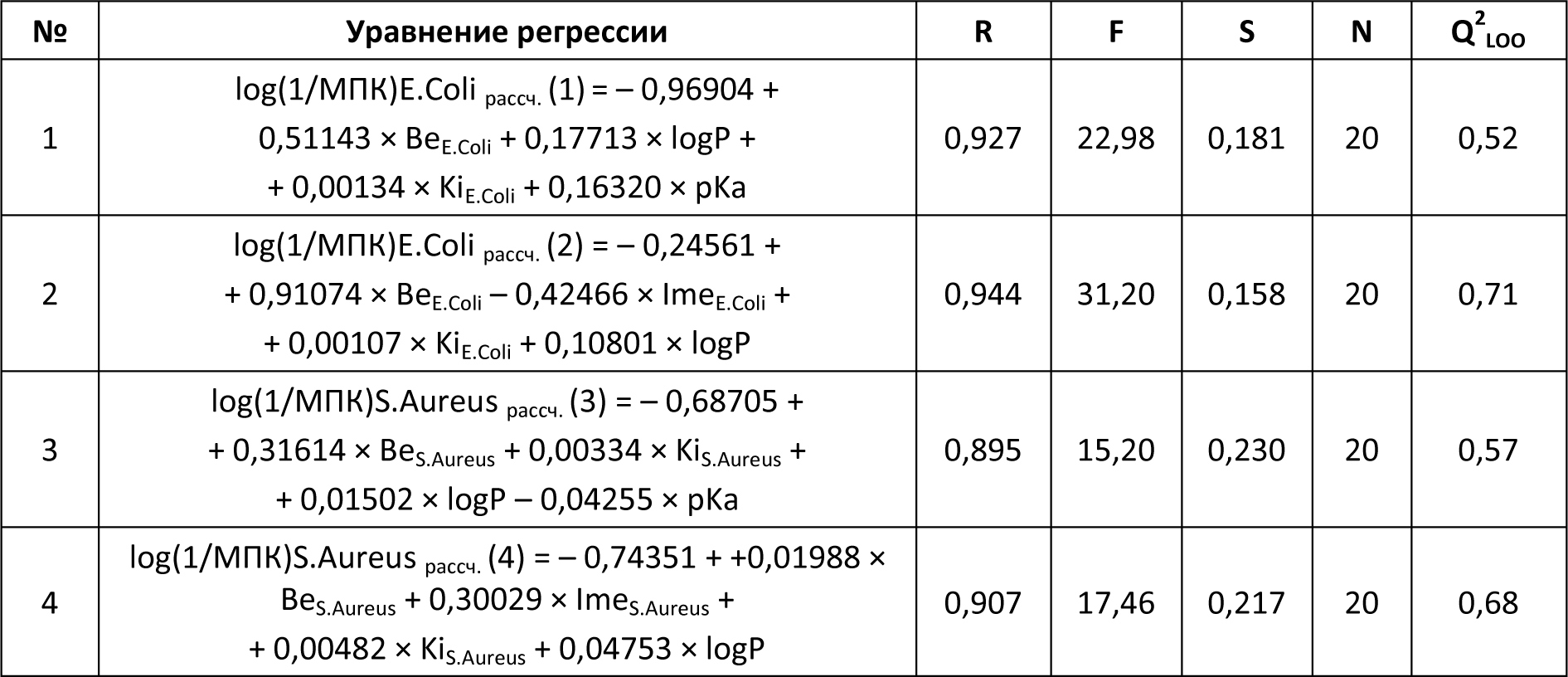 Проведена оценка составленных уравнений методом перекрестного контроля с выбором по одному (Leave-one-out cross-validation, LOO) (Q2LOO), с использованием программы Statographics. Определен коэффициент детерминации предсказаний Q2LOO, показывающий значимость составленных уравнений. По результатам проверки можно сделать вывод, что все составленные предсказательные модели на основе уравнений 1-4 являются значимыми, так как значение Q2 по LOO для надежных предсказательных моделей должно быть не менее 0,5 [19]. К наиболее значимым по рассчитанным критериям R, F, Q2LOO относятся модели в виде уравнений №2 и №4. Полученные уравнения апробированы для теоретического расчёта МПК E.Coli и S.Aureus на примере 4 соединений (21 – 24) из ряда замещенных амидов и гидразидов N-ароилантраниловых кислот (4 соединения). 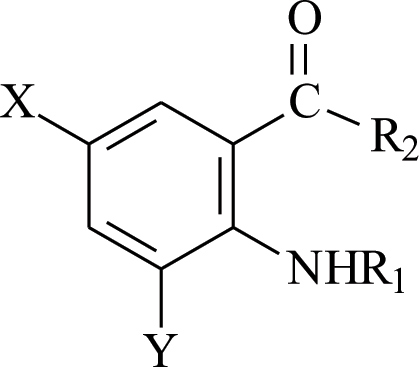 X=Br, Y=H, R1 = CO 2–COOH C6H4, R2 = NH C6H5 (21); X=Br, Y=H, R1 = CO 4–Cl C6H4, R2 = NH C6H5 (22); X=Br, Y=H, R1 = CO 2–COOHC6H4, R2 = NH2 (23); X=Br, Y=H, R1 = CO 2-фурил, R2 = NH2 (24). Вначале были проведены расчёты физико-химических дескрипторов (logPрассч и pKaрассч) и скоринговых функций (таблица 5) по ферментам гидролаза E.Coli (BeE.Coli, ImeE.Coli, KiE.Coli) и S.Aureus (BeS.Aureus, ImeS.Aureus, KiS.Aureus) методом молекулярного докинга программой AutoDock 4.0, и на их основе было рассчитано МПКрассч. (E.Coli) (таблица 6) и МПКрассч. (S.Aureus) (таблица 7). Затем была проведена проверка полученных результатов расчётов на экспериментальных данных, были определены МПКэксп. (E.Coli) и МПКэксп. (S.Aureus). Таблица 5. Скоринговые функции рассчитанные программой AutoDock 4.0 по E.Coli и S.Aureus, logPрассч, pKaрассч замещенных амидов и гидразидов N-ароилантраниловых кислот (4 соединения) 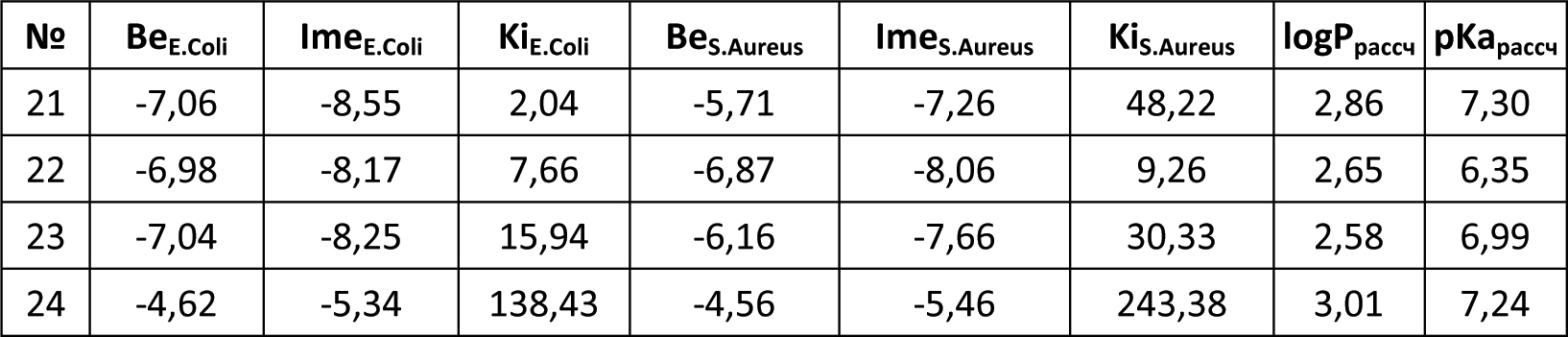 Таблица 6. log(1/МПК)E.Coli рассч., МПКрассч. (E.Coli), log(1/МПК)E.Coli эксп., МПКэксп. (E.Coli) замещенных амидов и гидразидов N-ароилантраниловых кислот (4 соединения) 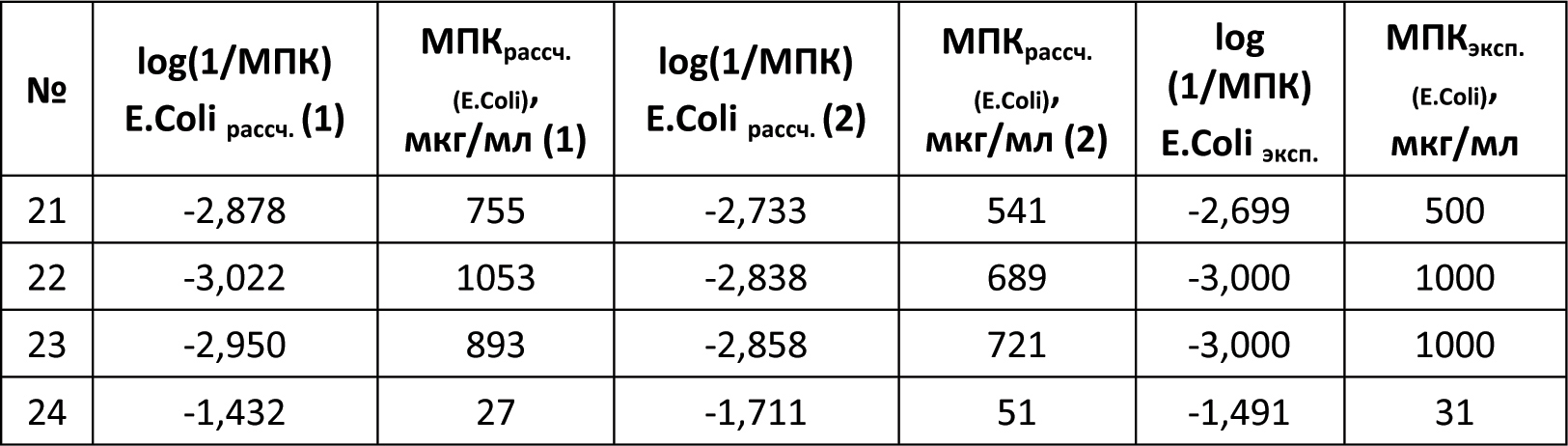 Таблица 7. log(1/МПК)S.Aureusрассч., МПКрассч. (S.Aureus), log(1/МПК)S.Aureus эксп., МПКэксп. (S.Aureus) замещенных амидов и гидразидов N-ароилантраниловых кислот (4 соединения) 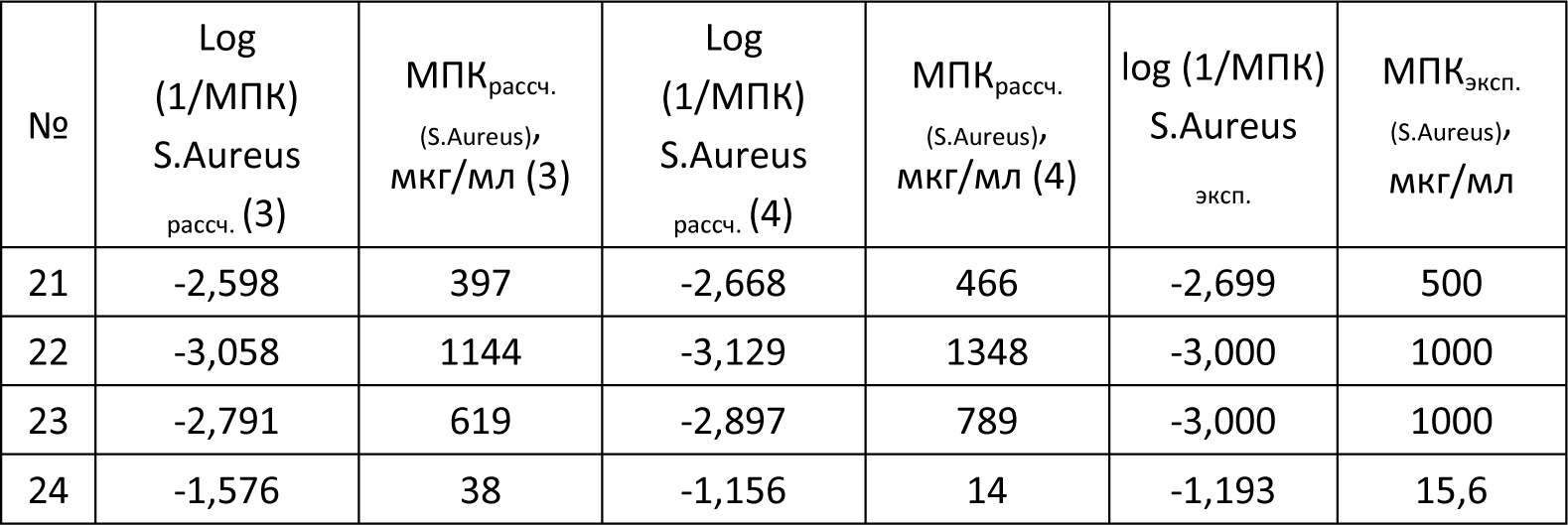 Результаты прогнозируемых значений МПКрассч. (E.Coli) и МПКрассч.(S.Aureus) и экспериментально найденных оказались близки (таблицы 6 и 7). Расчет прогнозируемой ПМА (МПКрассч.(E.Coli), МПКрассч.(S.Aureus), мкг/мл) для 4 соединений, показал, что экспериментально синтезировать целесообразно соединение 24, прогноз ПМА по уравнениям 1-4 составляет: МПКрассч. (E.Coli) (1) = 27 мкг/мл; МПКрассч.(E.Coli) (2) = 51 мкг/мл; МПКрассч. (S.Aureus) (3) = 38 мкг/мл; МПКрассч. (S.Aureus) (4) = 14 мкг/мл. Полученный результат прогноза ПМА подтверждается экспериментальным исследованием, в результате которого определено МПКэксп. соединения 24, которое составляет 31 мкг/мл по E.Coli и 15,6 мкг/мл по S.Aureus (таблицы 6 и 7). Для оценки качества прогноза ПМАрассч. (МПКрассч. (E.Coli) (1), МПКрассч. (E.Coli) (2), МПКрассч.(S.Aureus) (3), МПКрассч.(S.Aureus) (4), мкг/мл) составлены линейные уравнения взаимосвязи с экспериментальными значениями ПМАэксп. (МПКэксп.(E.Coli) и МПКэксп.(S.Aureus)): МПКрассч. (E.Coli) (1) = 0,92 × МПКэксп. (E.Coli) + 99,20; Rпрогн.= 0,945. МПКрассч. (E.Coli) (2) = 0,63 × МПКэксп. (E.Coli) + 95,87; Rпрогн.= 0,961. МПКрассч. (S.Aureus) (3) = 0,86 × МПКэксп. (S.Aureus) + 3,81; Rпрогн.= 0,884. МПКрассч. (S.Aureus) (4) = 1,08 × МПКэксп. (S.Aureus) – 27,55; Rпрогн.= 0,911. Линейные зависимости для уравнений 2 и 4, из четырех составленных, с коэффициентами корреляции (Rпрогн. (2)= 0,961 и Rпрогн. (4)= 0,911), показывают высокую степень связи ПМАрассч. c ПМАэксп.. Выводы Рассчитаны квантово-химические и физико-химические дескрипторы: константа липофильности (logPрассч), константа кислотности (pKaрассч) и основности (pKврассч). Установлено, что большинство из двадцати изученных замещенных амидов и гидразидов N-ароилантраниловых кислот обладает слабой бактериостатической активностью по отношению к E.Coli и S.Aureus (таблицы 2 и 3). Наиболее высокую бактериостатическую активность равную 15,6 мкг/мл, в отношении обоих штаммов, проявляет только соединение 2, по структуре амид тетрахлорфталил 5-бромантраниловой кислоты. Проведены докинг исследования анализируемых соединений, ряда замещенных амидов и гидразидов N-ароилантраниловых кислот методом молекулярного докинга программой AutoDock 4.0, в составе программного комплекса MGL Tools 1.5.6. При проведении молекулярного докинга использовали трёхмерные модели молекул фермента пептид деформилаза (гидролаза) E.Coli и S.Aureus. Выполнен множественный линейный регрессионный анализ, составлены 4 уравнения (таблица 4) зависимости экспериментально определенной ПМА (log(1/МПК)E.Coli и log(1/МПК)S.Aureus) от logPрассч, pKaрассч, BeE.Coli, ImeE.Coli, KiE.Coli, BeS.Aureus, ImeS.Aureus, KiS.Aureus. Проведена оценка составленных уравнений методом перекрестного контроля с выбором по одному (Leave-one-out cross-validation, LOO) (Q2LOO), с использованием программы Statographics. Определен коэффициент детерминации предсказаний Q2LOO, показывающий значимость составленных уравнений. Составленные уравнения апробированы для теоретического расчёта МПК E.Coli и S.Aureus на примере 4 соединений (21-24) из ряда замещенных амидов и гидразидов N-ароилантраниловых кислот. В результате проведенной проверки линейные зависимости для уравнений №2 и №4, из четырех составленных, с коэффициентами корреляции (Rпрогн. (2)= 0,961 и Rпрогн. (4)= 0,911), показывают высокую степень связи ПМАрассч. c ПМАэксп.. Предложены корреляционные уравнения №2 и №4, для проведения дальнейших исследований структура-противомикробная активность, которые позволяют производить достоверный и экономически выгодный прогноз уровня противомикробной активности, выявляя соединения с высокой ПМА до проведения их синтеза и биологических испытаний. Таким образом, можно сделать вывод, что получены уравнения регрессии взаимосвязи "структура-активность" с использованием метода молекулярного докинга и рассчитанных физико-химических дескрипторов, для поиска биологически активных соединений с противомикробной активностью в ряду производных антраниловой кислоты. Список литературы 1. Андрюков К.В., Коркодинова Л.М. Квантово-химические параметры в исследовании зависимости структура-ионизация N-замещенных моно (ди) галоген (Н) антраниловых кислот, их амидов и гидразидов. Химико-фармацевтический журнал 2016; 50 (3): 23-27. 2. Андрюков К.В., Коркодинова Л.М. Молекулярный докинг в изучении взаимодействия амидов и гидразидов N-ароилзамещенных алоген(Н)антраниловых кислот с циклооксигеназой 1, проявляющих противовоспалительную активность. Химико-фармацевтический журнал 2018; 52 (5): 29-32. 3. Андрюков К.В., Коркодинова Л.М. Прогнозирование коэффициента распределения октанол – вода производных N-арилзамещенных антраниловых кислот. Химико-фармацевтический журнал 2013; 47 (12): 38-41. 4. Андрюков К.В. Синтез, свойства и биологическая активность моно(ди)бромзамещенных производных антраниловой кислоты, изучение связи строения с действием с использованием констант ионизации и квантово-химических расчётов: Дисс. на соискание ученой степени к.фарм.н., Пермь, 2006. 166 с. 5. Андрюков К.В., Томилов М.В., Коркодинова Л.М., Одегова Т.Ф. Синтез и противомикробная активность замещенных амидов и гидразидов N-ацил-5-бромантраниловых кислот. Химико-фармацевтический журнал 2007; 41 (9): 29-31. 6. Подчезерцева А.В. Синтез, биологическая активность и установление количественных соотношений "структура-противовоспалительная активность" в ряду N-ацилантраниловых кислот и их амидов. Дисс. на соискание ученой степени к.фарм.н., Пермь, 2004. 142 с. 7. Руководство по экспериментальному (доклиническому) изучению новых фармакологических веществ. 2000. М., Бионт, 540. 8. Mohamed Eissa A.A., Soliman G.A., Khataibeh M.H. Design, Synthesis and Anti-inflammatory Activity of Structurally Simple Anthranilic Acid Congeners Devoid of Ulcerogenic Side Effects. Chemical and Pharmaceutical Bulletin 2012; 60 (10): 1290-1300. 9. Guilloteau J.P., Mathieu M., Giglione C., Blanc, V., Dupuy A., Chevrier M., Gil P., Famechon A., Meinnel T., Mikol V. Crystal structure of E.coli peptide deformylase complexed with antibiotic actinonin. J. Mol. Biol 2002; 320: 951-962. 10. Al-Rahawi K., Al-Kaf A., Shada Y., El-Nabtity S., El-Sayed K., Al-Shoba N. Synthesis and biological activities of 2-carboxyphenyloxamoylamino acids, their salts with 2-ethoxy-6,9-Diaminoacridine and D-glucosamine. Adv. Pharmacoepidem. Drug Safety 2013; 2 (2): 2-6. 11. Abouzid K.A., Khalil N.A., Ahmed E.M., Zaitone S.A. Synthesis and Biological Evaluation of New Heteroaryl Carboxylic Acid Derivatives as Anti-Inflammatory-Analgesic Agents. Chemical and Pharmaceutical Bulletin 2013; 61 (2): 222-228. 12. Kuldeep M., Pradeep K., Balasubramanian N., Synthesis, antimicrobial evaluation, ot-QSAR and mt-QSAR studies of 2-amino benzoic acid derivatives. Medicinal Chemistry Research 2012; 21 (3): 293-307. 13. Beniwal M., Lather V., Judge V., Jain N., Beniwal A. Anti-inflammatory, antimicrobial activity and QSAR studies of anthranilic acid derivatives. World journal of pharmacy and pharmaceutical sciences 2015; 4(09): 1443-1451. 14. Morris G.M., Huey R., Olson A.J. Using AutoDock for ligand-receptor docking. Curr. Protoc. Bioinformatics 2008; 11(13): 34-37. 15. El-Hashash M.A., Azab M.E., Faty R.A., Amr Ael-G. Synthesis, Antimicrobial and Anti-inflammatory Activity of Some New Benzoxazinone and Quinazolinone Candidates. Chemical and Pharmaceutical Bulletin 2016; 64 (3): 263-271. 16. Prudhomme M., Dauphin G., Guyot J., Jeminet G. Semisynthesis of A23187 (Calcimycin) analogs II. Introduction of a methyl group on the benzoxazole ring. The Journal of antibiotics 1984, 37 (6): 627-634. 17. Nadendla R.R., Mukkanti K., Sudhakar G. Rao. Microwave Synthesis of some new Quinazolinone Formazans for their Antimicrobial and Antihelminthic Activities. Current Trends in Biotechnology and Pharmacy 2010; 4 (1): 545-550. 18. Shahzad M., Jamshaid A. Synthesis, Antioxidant and Antimicrobial activity of 4-aminophenol and 2-aminobenzoic acid based novel azo compounds. Asian J. Chem. 2015; 27 (10): 3551-3554. 19. Sippl W., Contreras J.M., Parrot I., Rival Y.M., Wermuth C.G. Structure-based 3D QSAR and design of novel acetylcholinesterase inhibitors. Journal of Computer-Aided Molecular Design 2001; 15: 395-410. 20. Yoon H.J., Kim H.L., Lee S.K., Kim H.W. Crystal structure of peptide deformylase from Staphylococcus aureus in complex with actinonin, a naturally occurring antibacterial agent. Proteins 2004; 57: 639-642.
Corresponding Author: Andryukov Konstantin; e-mail: k_andrukov@mail.ru. Conflict of interest. None declared. Funding. The study had no sponsorship. Abstract The article focuses on the use of molecular docking for the enzyme E. Coli hydrolase (S. Aureus) in the study of the "structure-activity" relationship in the series of substituted amides and hydrazides of N-aroyl anthranilic acids. For the construction of the structure-activity model, we used the results of molecular docking on the enzyme peptide deformylase (hydrolase) E.Coli and S.Aureus of substituted amides and hydrazides of N-aroyl anthranilic acids. During the docking, E. Coli hydrolase enzymes (PDB ID code: 1LRU) and S. Aureus (PDB ID code: 1Q1Y) were used, the structures of which were obtained from the RCSB Protein Data Bank database. Before carrying out molecular docking, optimization and quantum-chemical calculation of the structures of the studied compounds was performed using the PM3 semiempirical method applying Gaussian 03 software. Significant parameters were determined: total electric field strength Σ (Е), potential Σ (φ) and the absolute value of charge Σ (|q|) on the atoms of oxygen, nitrogen, carbon and hydrogen. We carried out theoretical calculation of the physicochemical descriptors of the compounds under study: lipophilic constants (log Pcalc), acidity constants (pKacalc) and basicity (pKвcalc). Ligand-receptor interaction modeling was performed by the AutoDock 4.0 software as part of the MGL Tools 1.5.6 software package, using the Lamarkian genetic algorithm. As a result of docking for enzymes hydrolase E.Coli and S.Aureus, scoring functions were obtained: binding energy (Binding energy (BeE.Coli and BeS.Aureus)), intermolecular energy (Intermolecular energy (ImeE.Coli and ImeS.Aureus)) and inhibition constant (KiE.Coli and KiS.Aureus) characterizing the interaction of the ligand with the receptor. Studies of the dependence of antimicrobial activity (AA) from scoring functions and physicochemical descriptors were carried out. Structure-activity models were compiled by conducting multiple linear regression analysis using the Statistica 6 program. Four correlation equations were compiled relating the scoring functions and physico-chemical descriptors with antimicrobial activity using 20 compounds. A check was performed for the models created using the example of 4 compounds. Linear dependences of AAcalc. from experimental values of AAexp. (MICexp.(E.Coli) and MICexp.(S.Aureus) for equations 2 and 4, made up of four, with correlation coefficients (Rpred. (2) = 0.961 and Rpred. (4) = 0.911), show a high degree of communication AAcalc. with AAexp.. Key words amide, hydrazide, аnthranilic acid, molecular docking, antimicrobial activity, quantum chemical parameters, structure-activity References 1. Andryukov K.V., Korkodinova L.M. Kvantovo-himicheskie parametry v issledovanii zavisimosti struktura-ionizaciya N-zameshchennyh mono (di)galogen(n) antranilovyh kislot, ih amidov i gidrazidov [Quantum and chemical parameters in a dependence research structure-ionization of the anthranilic acids N-replaced mono halogen (H), their amides and hydrazides]. Himiko-farmacevticheskij zhurnal [Chemical and Pharmaceutical Journal] 2016; 50 (3): 23-27. (In Russ.) 2. Andryukov K.V., Korkodinova L.M. Molekulyarnyj doking v izuchenii vzaimodejstviya amidov i gidrazidov N-aroilzameshchennyh alogen(N)antranilovyh kislot s ciklooksigenazoj 1, proyavlyayushchih protivovospalitel'nuyu aktivnost' [Molecular docking study of the interaction of cyclooxygenase 1 with N-aroyl-substituted halogen(H)anthranilic acid amides and hydrazides exhibiting anti-inflammatory activity]. Himiko-farmacevticheskij zhurnal [Chemical and Pharmaceutical Journal] 2018; 52 (5): 29-32. (In Russ.) 3. Andryukov K.V., Korkodinova L.M. Prognozirovanie koehfficienta raspredeleniya oktanol-voda proizvodnyh N-arilzameshchennyh antranilovyh kislot [Prediction of the octanol-water partition coefficients of N-aryl-substituted anthranilic acid derivatives]. Himiko-farmacevticheskij zhurnal [Chemical and Pharmaceutical Journal] 2013; 47 (12): 38-41. (In Russ.) 4. Andryukov K.V. Sintez, svojstva i biologicheskaya aktivnost' mono(di)bromzameshchennyh proizvodnyh antranilovoj kisloty, izuchenie svyazi stroeniya s dejstviem s ispol'zovaniem konstant ionizacii i kvantovo-himicheskih raschyotov [Synthesis, properties and biological activity of mono(di)bromosubstituted anthranilic acid derivatives, study of structure-activity relationship using ionization constants and quantum chemical calculations]. Diss. na soiskanie uchenoj stepeni k.farm.n. [PhD (Pharmacy) Thesis]. Perm', 2006. (In Russ.) 5. Andryukov K.V., Tomilov M.V., Korkodinova L.M., Odegova T.F. Sintez i protivomikrobnaya aktivnost' zameshchennyh amidov i gidrazidov N-acil-5-bromantranilovyh kislot [Synthesis and antimicrobial activity of substituted amides and hydrazides of N-acyl-5-bromanthranilic acids]. Himiko-farmacevticheskij zhurnal [Chemical and Pharmaceutical Journal] 2007; 41 (9): 29-31. (In Russ.) 6. Podchezerceva A.V. Sintez, biologicheskaya aktivnost' i ustanovlenie kolichestvennyh sootnoshenij "struktura-protivovospalitel'naya aktivnost'" v ryadu N-acilantranilovyh kislot i ih amidov [Synthesis, biological activity and the establishment of quantitative correlations "structure-anti-inflammatory activity" in the row N-acylanthranilic acids and their amides]. Diss. na soiskanie uchenoj stepeni k.farm.n. [PhD (Pharmacy) Thesis]. Perm', 2004. (In Russ.) 7. Rukovodstvo po ehksperimental'nomu (doklinicheskomu) izucheniyu novyh farmakologicheskih veshchestv [Guidelines for experimental (preclinical) study of new pharmacological substances]. 2000. Moscow, Biont. (In Russ.) 8. Mohamed Eissa A.A., Soliman G.A., Khataibeh M.H. Design, Synthesis and Anti-inflammatory Activity of Structurally Simple Anthranilic Acid Congeners Devoid of Ulcerogenic Side Effects. Chemical and Pharmaceutical Bulletin 2012; 60 (10): 1290-1300. 9. Guilloteau J.P., Mathieu M., Giglione C., Blanc, V., Dupuy A., Chevrier M., Gil P., Famechon A., Meinnel T., Mikol V. Crystal structure of E.coli peptide deformylase complexed with antibiotic actinonin. J. Mol. Biol 2002; 320: 951-962. 10. Al-Rahawi K., Al-Kaf A., Shada Y., El-Nabtity S., El-Sayed K., Al-Shoba N. Synthesis and biological activities of 2-carboxyphenyloxamoylamino acids, their salts with 2-ethoxy-6,9-Diaminoacridine and D-glucosamine. Adv. Pharmacoepidem. Drug Safety 2013; 2 (2): 2-6. 11. Abouzid K.A., Khalil N.A., Ahmed E.M., Zaitone S.A. Synthesis and Biological Evaluation of New Heteroaryl Carboxylic Acid Derivatives as Anti-Inflammatory-Analgesic Agents. Chemical and Pharmaceutical Bulletin 2013; 61 (2): 222-228. 12. Kuldeep M., Pradeep K., Balasubramanian N., Synthesis, antimicrobial evaluation, ot-QSAR and mt-QSAR studies of 2-amino benzoic acid derivatives. Medicinal Chemistry Research 2012; 21 (3): 293-307. 13. Beniwal M., Lather V., Judge V., Jain N., Beniwal A. Anti-inflammatory, antimicrobial activity and QSAR studies of anthranilic acid derivatives. World journal of pharmacy and pharmaceutical sciences 2015; 4(09): 1443-1451. 14. Morris G.M., Huey R., Olson A.J. Using AutoDock for ligand-receptor docking. Curr. Protoc. Bioinformatics 2008; 11(13): 34-37. 15. El-Hashash M.A., Azab M.E., Faty R.A., Amr Ael-G. Synthesis, Antimicrobial and Anti-inflammatory Activity of Some New Benzoxazinone and Quinazolinone Candidates. Chemical and Pharmaceutical Bulletin 2016; 64 (3): 263-271. 16. Prudhomme M., Dauphin G., Guyot J., Jeminet G. Semisynthesis of A23187 (Calcimycin) analogs II. Introduction of a methyl group on the benzoxazole ring. The Journal of antibiotics 1984, 37 (6): 627-634. 17. Nadendla R.R., Mukkanti K., Sudhakar G. Rao. Microwave Synthesis of some new Quinazolinone Formazans for their Antimicrobial and Antihelminthic Activities. Current Trends in Biotechnology and Pharmacy 2010; 4 (1): 545-550. 18. Shahzad M., Jamshaid A. Synthesis, Antioxidant and Antimicrobial activity of 4-aminophenol and 2-aminobenzoic acid based novel azo compounds. Asian J. Chem. 2015; 27 (10): 3551-3554. 19. Sippl W., Contreras J.M., Parrot I., Rival Y.M., Wermuth C.G. Structure-based 3D QSAR and design of novel acetylcholinesterase inhibitors. Journal of Computer-Aided Molecular Design 2001; 15: 395-410. 20. Yoon H.J., Kim H.L., Lee S.K., Kim H.W. Crystal structure of peptide deformylase from Staphylococcus aureus in complex with actinonin, a naturally occurring antibacterial agent. Proteins 2004; 57: 639-642. |
[ См. также ] Рубрики |
||||
|
|
|
Журнал «Медицина» © ООО "Инновационные социальные проекты"
|